O Nobel de Química vai para os desenvolvedores do AlphaFold AI que prevê estruturas de proteínas

David Baker, Demis Hassabis e John Jumper (da esquerda para a direita) ganharam o Nobel de Química pelo progresso de ferramentas computacionais que podem prever e projetar estruturas de proteínas.Crédito: Fundação BBVA
Pela primeira vez — e provavelmente não a última — um avanço científico possibilitado pela inteligência artificial (IA) foi reconhecido com um prémio Nobel. O Nobel de Química de 2024 foi concedido a John Jumper e Demis Hassabis do Google DeepMind em Londres, por desenvolverem uma ferramenta de IA revolucionária para prever estruturas de proteínas chamada AlphaFold, e David Baker, da Universidade de Washington em Seattle, por seu trabalho em computação. design de proteína, que foi reforçado por Al nos últimos anos.
“Espero que, quando olharmos para o AlphaFold, ele seja a primeira prova do incrível potencial da IA para acelerar a descoberta científica”, disse Hassabis em uma coletiva de imprensa na DeepMind em 9 de outubro. “É tão irreal neste momento.”

O que vem por aí para AlphaFold e a revolução do dobramento de proteínas AI
O impacto do AlphaFold, que foi revelado há apenas alguns anos, foi nada menos que transformador. A ferramenta disponibilizou estruturas proteicas – muitas vezes, mas nem sempre, altamente precisas – aos investigadores com o toque de um botão, e permitiu experiências que eram inimagináveis há uma década. “É uma grande revolução”, diz Christine Orengo, bióloga computacional da University College London, cujo laboratório utilizou estruturas previstas pelo AlphaFold para descobrir novas proteínas.
“Há muito que é um sonho aprender a prever a estrutura tridimensional das proteínas a partir do conhecimento das suas sequências de aminoácidos… durante várias décadas, isto foi considerado impossível”, disse o presidente do comité do Nobel, Heiner Linke, que investiga nanociência na Universidade de Lund, na Suécia. , durante o anúncio do prêmio. Os laureados deste ano “decifraram o código”, acrescentou. Os três vencedores dividem um prêmio de 11 milhões de coroas suecas (US$ 1 milhão).
IA premiada
DeepMind estreou AlphaFold em 2018, quando ganhou um concurso bienal de previsão de estrutura de proteínas chamado Avaliação Crítica de Previsão de Estrutura de Proteínas (CASP). Mas foi a segunda iteração da rede neural de aprendizagem profunda, revelada no final de 2020, que realmente abalou as ciências da vida. Muitas das previsões do AlphaFold2 no CASP foram tão precisas que eram indistinguíveis das estruturas proteicas resolvidas experimentalmente.
Hassabis, cofundador e executivo-chefe da DeepMind, e Jumper, chefe da equipe AlphaFold, lideraram o progresso do AlphaFold2. Para prever estruturas proteicas, a rede neural incorpora dados de bibliotecas de centenas de milhares de estruturas e milhões de sequências de proteínas relacionadas – que contêm informações sobre suas formas.

Como ganhar um prêmio Nobel: que tipo de cientista ganha medalhas?
Em particular, o sucesso do AlphaFold se deve em grande parte ao Protein Data Bank, um repositório disponível gratuitamente com mais de 200.000 estruturas de proteínas determinadas usando métodos que incluem cristolagrafia de raios X e microscopia crioeletrônica. “É uma lição de humildade cada vez que treinamos [AlphaFold] em anos de esforço. Cada ponto de dados representa anos de esforço de alguém”, disse Jumper na coletiva de imprensa da DeepMind.
Em 2021, a DeepMind disponibilizou gratuitamente o código subjacente do AlphaFold2, juntamente com os dados necessários para treinar o modelo. Uma base de dados AlphaFold, criada com o Instituto Europeu de Bioinformática do Laboratório Europeu de Biologia Molecular em Hinxton, Reino Unido, contém agora as estruturas de quase todas as proteínas de todos os organismos representados em bases de dados genéticas, cerca de 214 milhões de previsões no total. Este ano, a empresa lançou uma terceira versão do AlphaFold, que pode modelar outras moléculas que interagem com proteínas, como medicamentos.
A revolução que Jumper, Hassabis e os seus colegas desencadearam ainda está nos seus primórdios, e o impacto total do AlphaFold na ciência poderá não ser conhecido durante anos. A ferramenta já está ajudando os cientistas a obter novos insights.
Uma equipa pioneira utilizou a ferramenta, juntamente com dados experimentais, para mapear o complexo de poros nucleares, uma das maiores máquinas das nossas células que transporta moléculas para dentro e para fora do núcleo. No ano passado, duas equipes exploraram todo o banco de dados AlphaFold para descobrir os cantos mais obscuros do universo das proteínas, identificando novas famílias de proteínas e dobras e conexões surpreendentes na maquinaria da vida.
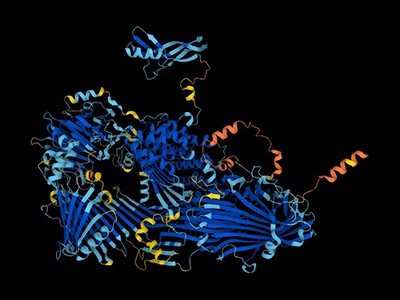
‘Todo o universo das proteínas’: a IA prevê a forma de quase todas as proteínas conhecidas
Muitos investigadores esperam que o AlphaFold e outras ferramentas de IA que ele inspirou transformem a medicina, mas ainda não está claro como, ou mesmo se, o AlphaFold irá agilizar o processo dispendioso e de múltiplas etapas de progresso de medicamentos seguros. Os cientistas que preparam as bases para novas vacinas estão achando o AlphaFold incrivelmente útil e, em alguns casos, revolucionário. Mas o AlphaFold é um complemento aos estudos experimentais e outras abordagens para mapear e ajustar a estrutura das proteínas virais para uso em vacinas.
Para a maioria dos investigadores, uma estrutura prevista é o início de um estudo, não o fim, diz Jan Kosinski, modelador estrutural do Laboratório Europeu de Biologia Molecular (EMBL) em Hamburgo, Alemanha. “No início havia esse medo de que ela substituísse a biologia estrutural, as pessoas perdessem empregos e daquela forma por diante. Na verdade, aconteceu exatamente o oposto”, acrescenta.
David Jones, bioinformático da University College London que colaborou com DeepMind na primeira versão do AlphaFold a partir de 2016, diz que um dos maiores impactos da ferramenta foi uma mudança na mentalidade do biólogo, “para dizer que os computadores são coisas que podem produzir hipóteses úteis que pode ser testado em laboratório”.
Criando novas proteínas
Mais de duas décadas antes de DeepMind começar a trabalhar no AlphaFold, o biofísico computacional David Baker e seus colegas desenvolveram uma ferramenta de software chamada Rosetta que modelava estruturas de proteínas usando princípios físicos. A ferramenta compara pequenos fragmentos de múltiplas estruturas e sequências proteicas existentes para identificar uma sequência proteica que pode se dobrar em um formato específico.
Inicialmente, o Rosetta foi aplicado à previsão de estruturas de proteínas – esteve entre as principais entradas em vários CASPs, antes do domínio do AlphaFold. Mas Baker logo percebeu que o modelo poderia ser invertido para projetar proteínas inteiramente novas.
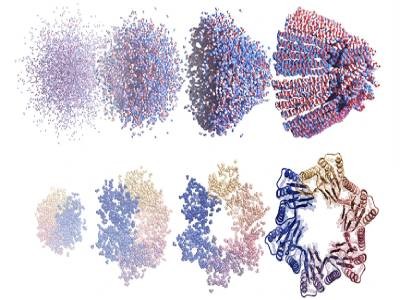
Ferramentas de IA estão projetando proteínas inteiramente novas que poderiam transformar a medicina
A ferramenta teve sucesso inicial na concepção de novas proteínas, incluindo novos tipos de enzimas, proteínas que se podem ligar firmemente a outras moléculas e nanopartículas de proteínas auto-montáveis que se assemelham a vírus (uma delas serviu de base para uma vacina contra a COVID-19 aprovada).
Quando o AlphaFold2 foi anunciado – mas ainda não lançado – Baker e sua equipe, incluindo o químico computacional Minkyung Baek, agora na Universidade Nacional de Seul, na Coreia do Sul, decidiram entender o software e aplicar alguns de seus truques a uma versão anterior baseada em IA do Roseta. A primeira versão da rede RoseTTAFold resultante teve um desempenho quase tão bom quanto o AlphaFold2. Desde 2021, ambas as redes têm sido continuamente melhoradas pelos seus desenvolvedores e outros cientistas para enfrentar novos desafios, como prever a estrutura de complexos de múltiplas proteínas diferentes que interagem.
Nos últimos anos, a equipe de Baker tem sido especialmente prolífica na aplicação do aprendizado de máquina à razão de ser de seu laboratório: criar novas proteínas nunca vistas antes na natureza. Uma ferramenta desenvolvida recentemente pela equipe de Baker que combina RoseTTAFold com redes neurais de difusão geradoras de imagens levou a uma mudança radical na capacidade dos pesquisadores de projetar proteínas.
Progresso rápido
Essas ferramentas têm sido um enorme acelerador e democratizador, diz Sergey Ovchinnikov, biólogo evolucionista do Instituto de Tecnologia de Massachusetts, em Cambridge, que fez o seu doutoramento no laboratório de Baker. Costumava levar semanas para o Rosetta rodar em centenas de processadores para chegar a um design de proteína, uma tarefa que as ferramentas mais recentes baseadas em IA podem realizar em segundos. “Agora todos no mundo podem projetar proteínas”, diz ele.
“Fui profundamente inspirado por outros profissionais da área e pelas pessoas com quem trabalhei”, disse Baker, falando por telefone no anúncio do prêmio Nobel. “Eu estava sobre ombros de gigantes.”

Nobel de Medicina premiado por 'microRNAs' reguladores de genes
Martin Steinegger, biólogo computacional da Universidade Nacional de Seul, na Coreia do Sul, compara o impacto do AlphaFold, RoseTTAFold e outras ferramentas biológicas de IA ao das missões Apollo Moon, mostrando o que a engenharia pode alcançar. “Este é um momento semelhante para a previsão de estruturas e para o campo da biologia estrutural – apenas para ver o que é viável”, diz ele.
Poucos ficaram surpresos com a decisão do comitê do Nobel. Para Baker, “a maioria das pessoas pensava que era uma situação do tipo 'quando não se', pela quantidade de trabalho que ele realizou nessa área”, diz Jones. Jumper, ciente de que ele e Hassabis estavam nas listas de muitas pessoas, disse na coletiva de imprensa que não conseguiu dormir na noite anterior ao anúncio de hoje.
Para Jumper, as estruturas previstas que o AlphaFold oferece criam novas oportunidades para descobertas científicas. Milhões de cientistas já utilizaram as ferramentas e ele espera que não demore muito até que um deles receba uma chamada da Suécia. “O momento em que ficarei quase tão animado quanto este será o Prêmio Nobel que fala sobre o trabalho feito com AlphaFold”, disse ele.
